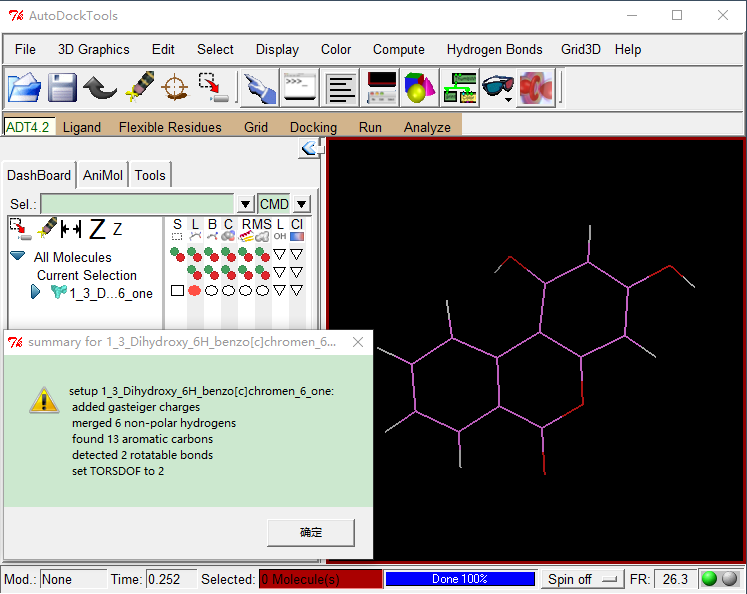
复现A Small-Molecule Compound Has Anti-influenza A Virus Activity by Acting as a “PB2 Inhibitor”中的蛋白质-配体结合图
软件准备
Chem3D
付费软件,自行购买安装MGLTools
MGLTools选择合适的版本下载安装,安装完成会出现AutoDockTools及其它三个图标AutoDock Vina
AutoDock Vina选择合适的版本下载安装,安装目录添加到环境变量开源版PyMOL
参考教程Windows安装免费开源PyMOL的过程安装Get Box插件
GetBox-PyMOL-Plugin下载安装center_of_mass.py
参考零基础PyMOL作图教程——绘制蛋白质-分子结合模式图中相应内容安装
分子对接
蛋白质文件准备
PyMOL命令行输入
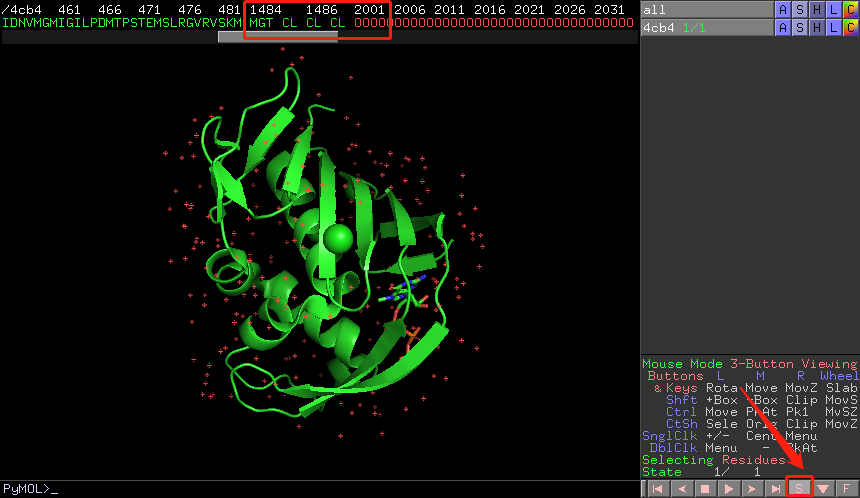
fetch 4cb4,type=pdb,回车,获得蛋白的pdb文件

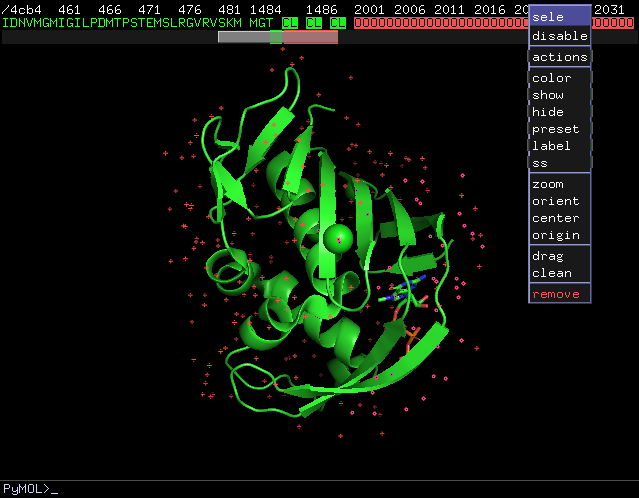
点击右下角S显示序列,MGT为共结晶的配体,CL为氯原子,O为水鼠标拖选序列中的氯原子和水,右键
remove,只保留蛋白和配体

菜单栏
File -> Export Molecule...,保存为4cb4_ligand.pdb,后续用于检测对接盒子同样的方式再把配体删除,只保留蛋白,保存为
4cb4_clean.pdb
AutoDockTools菜单栏
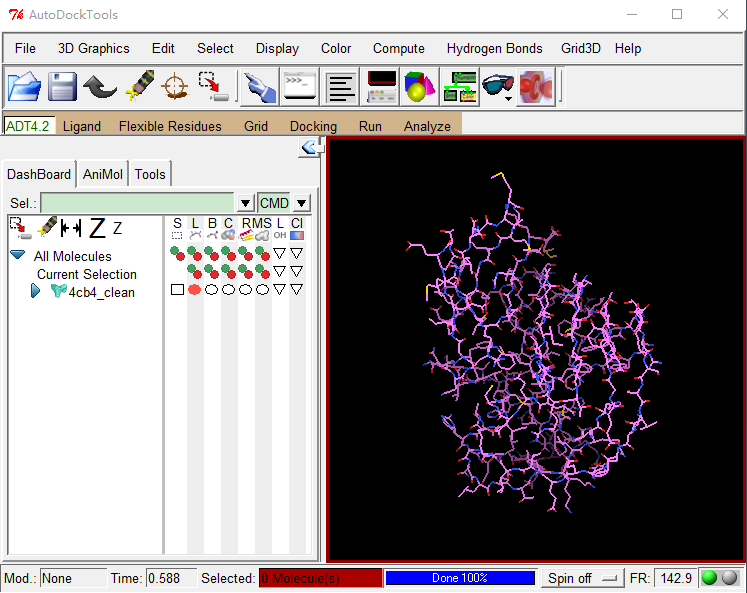
File -> Read Molecule打开4cb4_clean.pdb

加氢:
Edit -> Hydrogens -> Add -> OK

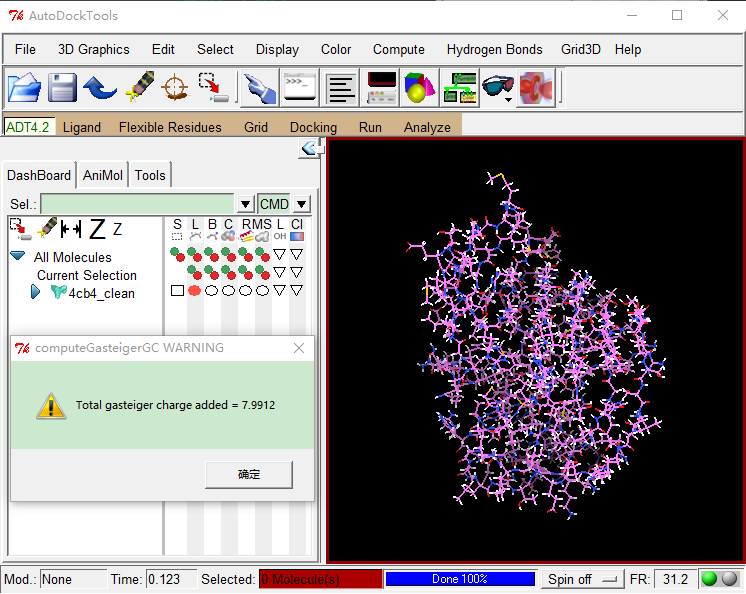
计算电荷:
Edit -> Charges -> Compute Gasteiger

添加原子类型:
Edit -> Atoms -> Assign AD4 type保存为
4cb4_clean.pdbqt:File -> Save -> Write PDBQT
配体文件准备
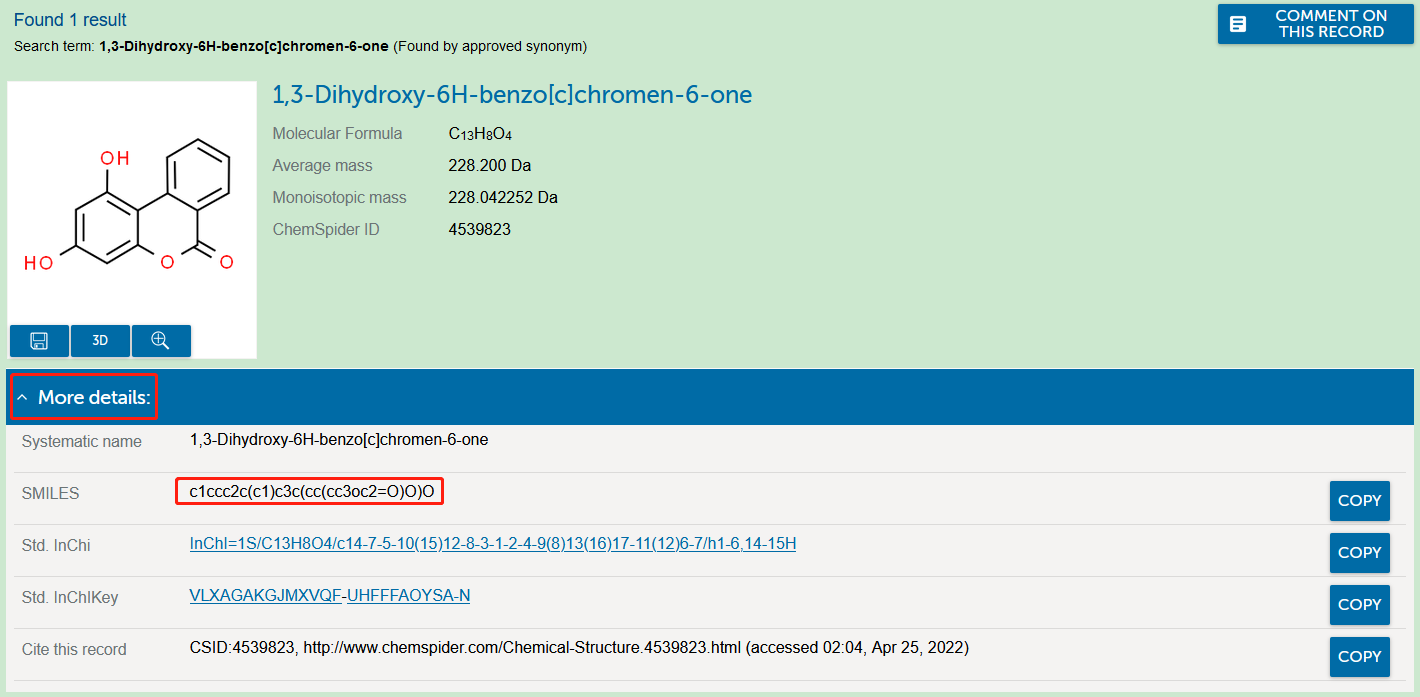
ChemSpider输入
1,3-Dihydroxy-6H-benzo[c]chromen-6-one搜索,搜索结果页点击More details展开,复制SMILES

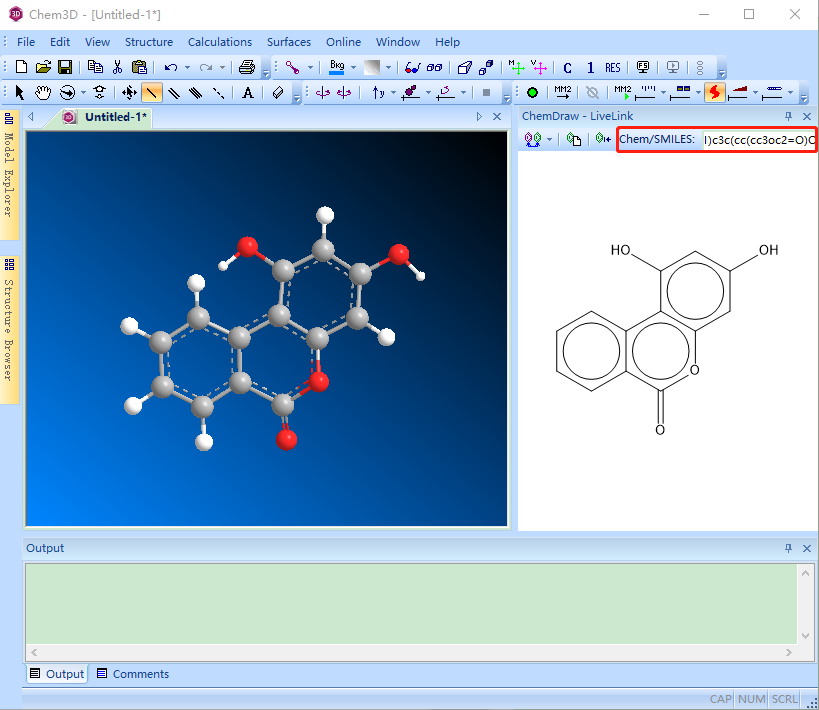
打开Chem3D,侧边栏输入复制的
SMILES,回车

力场能量最小化:
Calculations -> MMFF94 -> Perform MMFF94 Minimization保存为
1,3-Dihydroxy-6H-benzo[c]chromen-6-one.mol2
AutoDockTools
Ligand -> Input -> Open导入1,3-Dihydroxy-6H-benzo[c]chromen-6-one.mol2

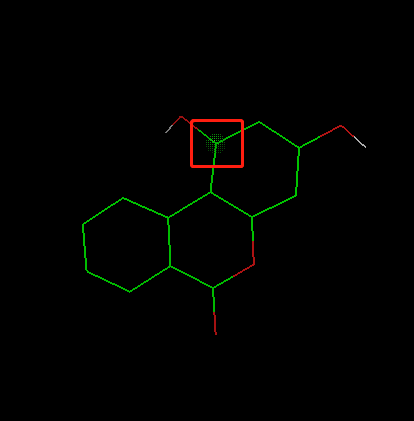
判定配体的root:
Ligand -> Torsion Tree -> Detect Root

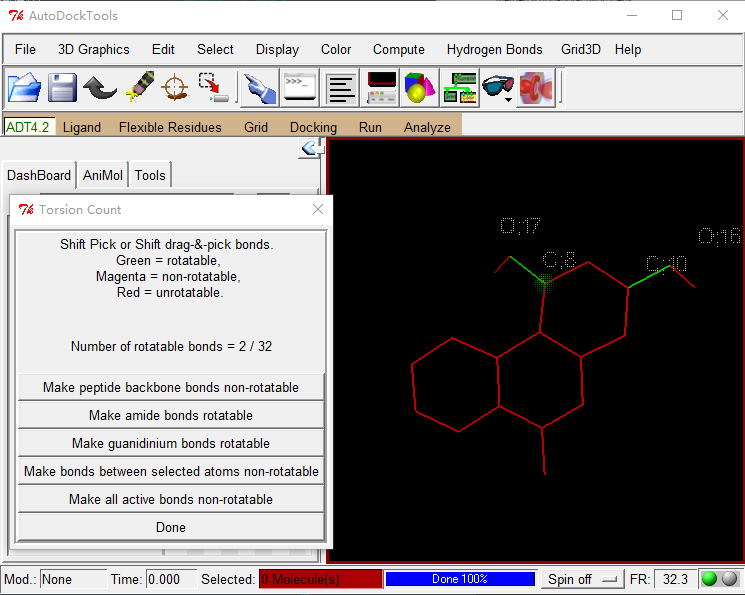
选择配体可扭转的键:
Ligand -> Torsion Tree -> Choose Torsions -> Done

保存为
1_3_Dihydroxy_6H_benzo[c]chromen_6_one.pdbqt:Ligand -> Output -> Save as PDBQT
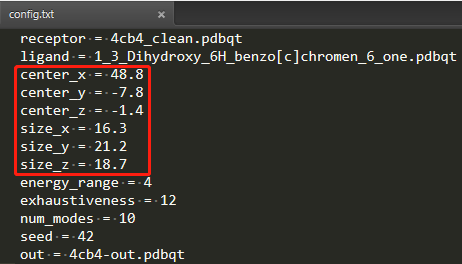
配置文件
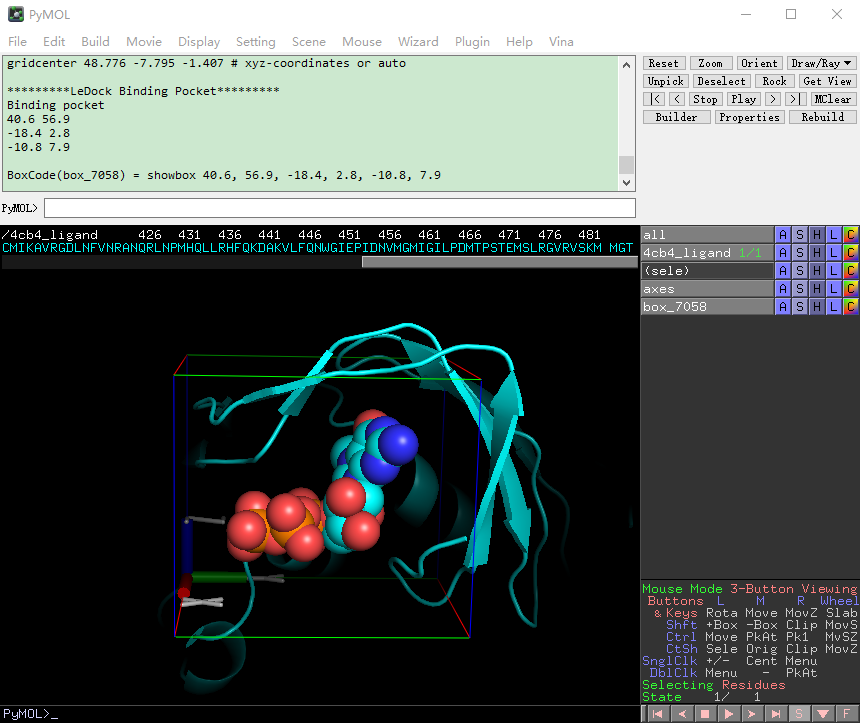
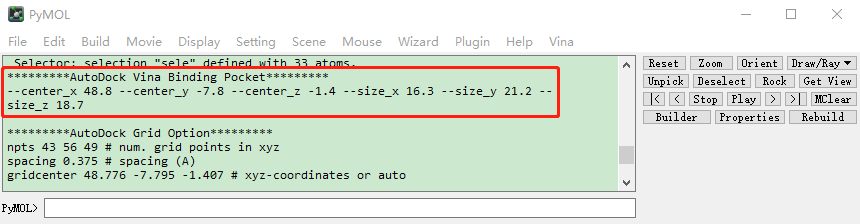
确定对接盒子
PyMOL打开
4cb4_ligand.pdb,鼠标点击序列中的MGT选中它,菜单栏Plugin -> Legacy Plugins -> GetBox Plugin -> Get box from selection (sele)

找到
AutoDock Vina Binding Pocket

创建配置文件
config.txt

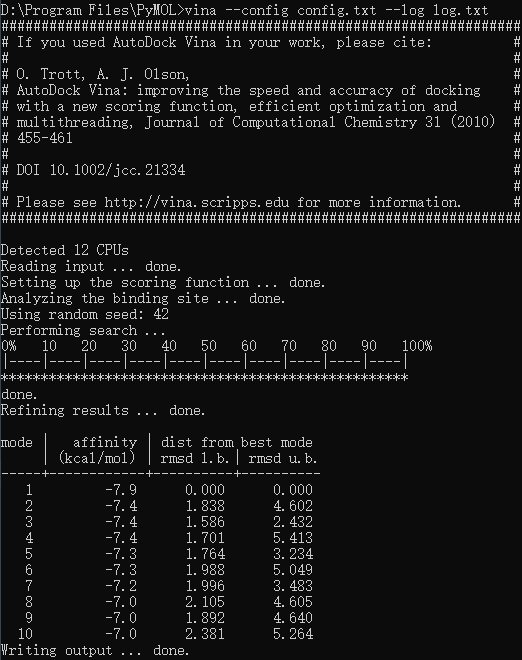
运行对接
打开命令行,切换到保存
receptor,ligand及配置文件的目录,运行vina --config config.txt --log log.txt

PyMOL绘图
主要参考高质量PyMOL作图教程
对接结果预处理
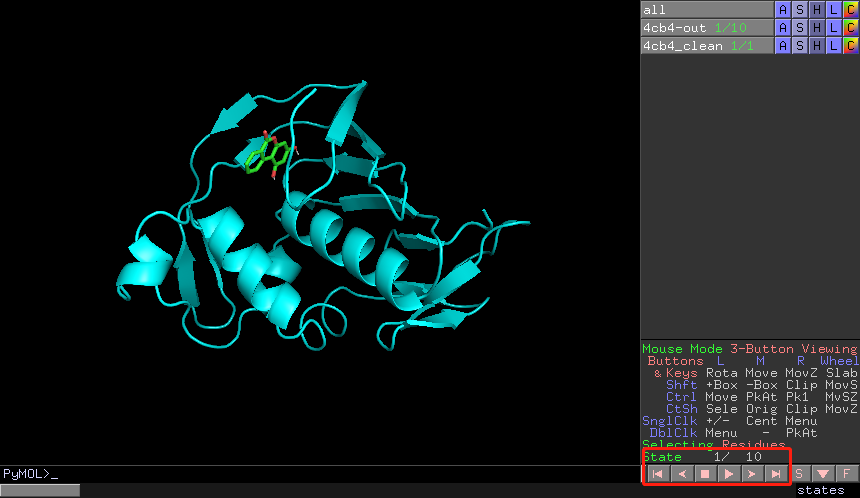
PyMOL打开
4cb4-out.pdbqt和4cb4_clean.pdb。4cb4-out.pdbqt有10个构象,可以通过右下方的方向键切换查看

切换到得分最高的第1个构象,菜单栏
File -> Export Molecule...,保存为4cb4_docked.pdb菜单栏
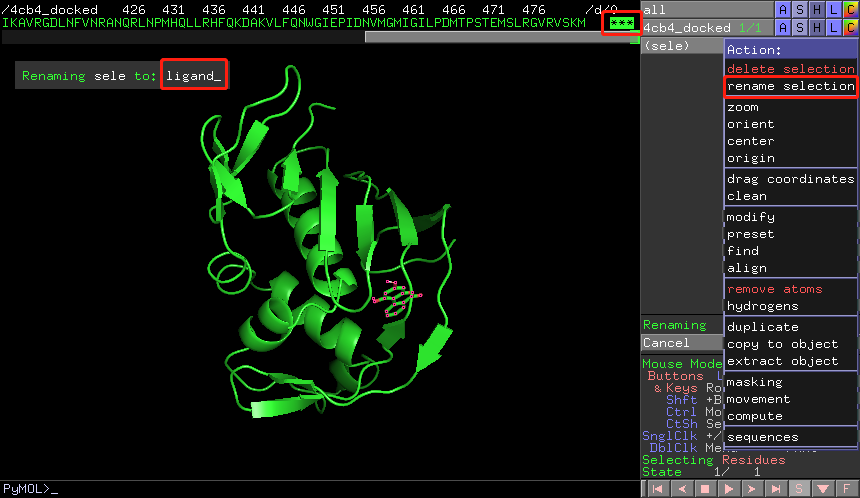
File -> Reinitialize -> Everything清空所有对象,重新打开4cb4_clean.pdb将配体重命名,选中序列中的
***,A -> rename selection,键盘输入ligand,回车

PyMOL命令行输入
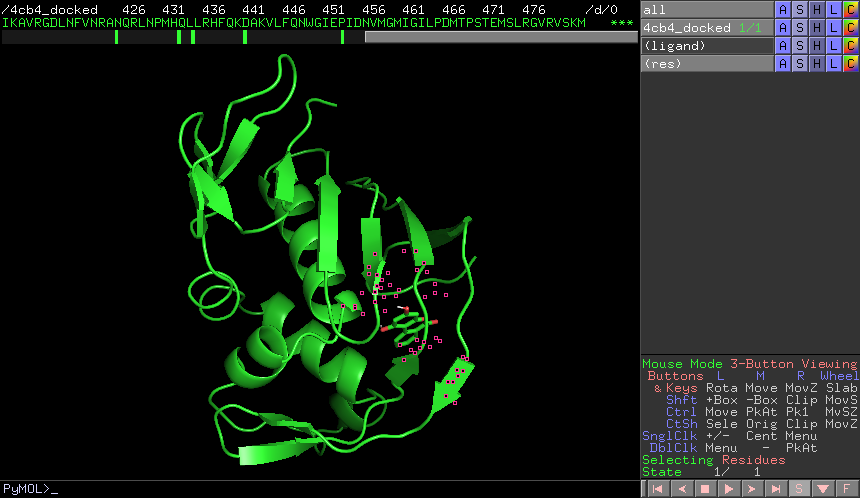
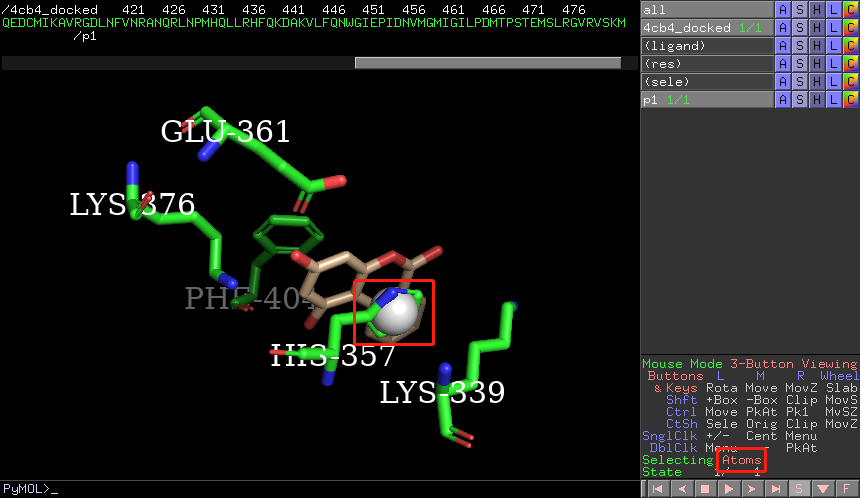
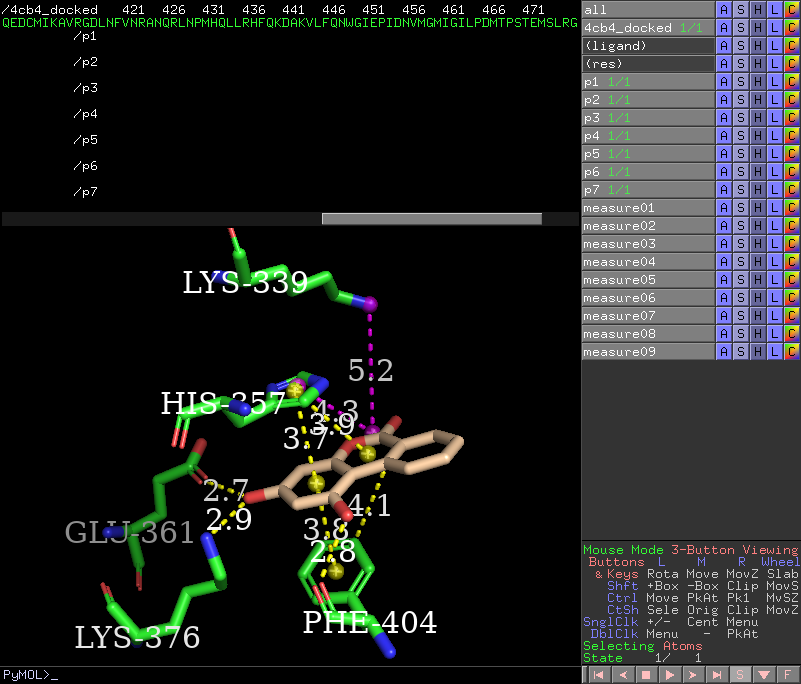
select res, resi 339+357+361+376+404,选中关键残基,命名为res

PyMOL命令行输入
hide,隐藏所有对象PyMOL命令行输入
show sticks, ligand | res,显示配体和关键残基的棍棒结构PyMOL命令行输入
hide sticks, h.,隐藏氢原子PyMOL命令行输入
zoom ligand | res,居中放大显示配体和关键残基PyMOL命令行输入
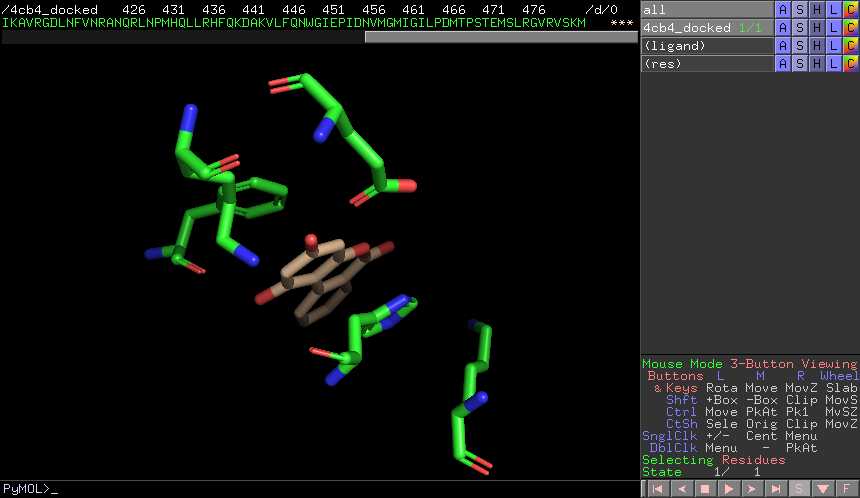
color wheat, ligand & name C*,将配体中的碳原子改为小麦色

PyMOL命令行输入
set label_size, 30,设置字体大小为30磅PyMOL命令行输入
set label_font_id, 9,设为9号字体右侧
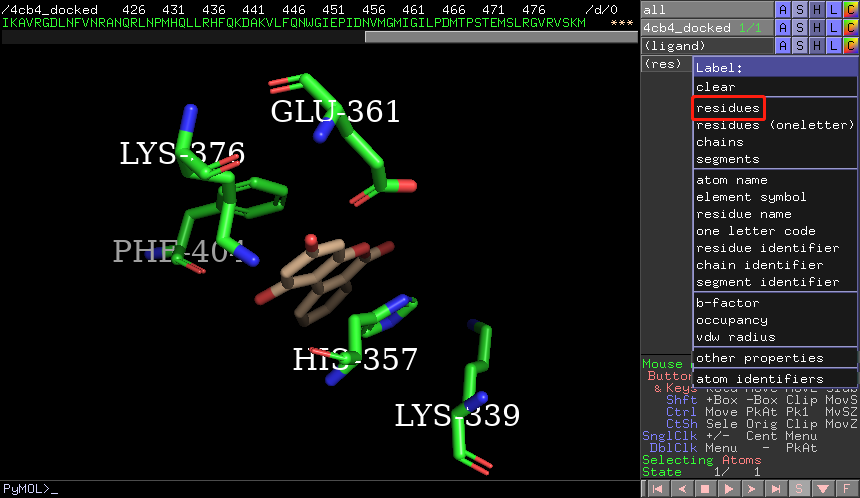
(res)点击L -> residues显示关键残基标签

绘制相互作用力
PyMOL命令行输入
import center_of_mass导入插件右下角选择模式改为
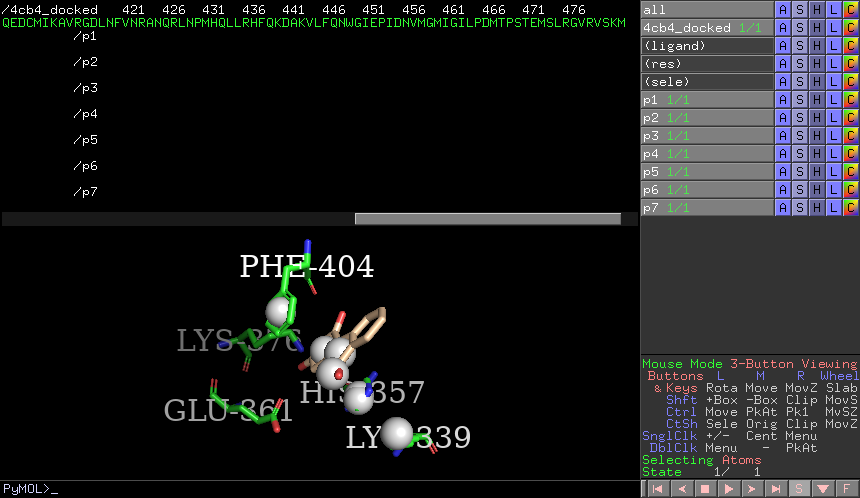
Atom,点击选中HIS-357咪唑环上的5个碳原子,命令行输入com sele, object=p1,在选中的原子的几何中心创建小球,命名为p1

点击选中配体间二羟基苯环上任意对位的2个碳原子或全部6个碳原子,命令行输入
com sele, object=p2点击选中配体吡喃酮环上任意对位的2个原子或全部6个原子,命令行输入
com sele, object=p3点击选中
PHE-404苯环上任意对位的2个碳原子或全部6个碳原子,命令行输入com sele, object=p4点击选中配体酯基的2个氧原子,命令行输入
com sele, object=p5点击选中
LYS-339的氮原子,命令行输入com sele, object=p6点击选中
HIS-357咪唑基的2个氮原子,命令行输入com sele, object=p7

PyMOL命令行输入
set sphere_scale, 0.35设置球的大小PyMOL命令行输入
set sphere_transparency, 0.5设置球的透明度PyMOL命令行输入
set sphere_color, yellow将球设为黄色PyMOL命令行输入
set sphere_color, magenta, p5 | p6 | p7将p5,p6,p7三个小球设为洋红色

菜单栏
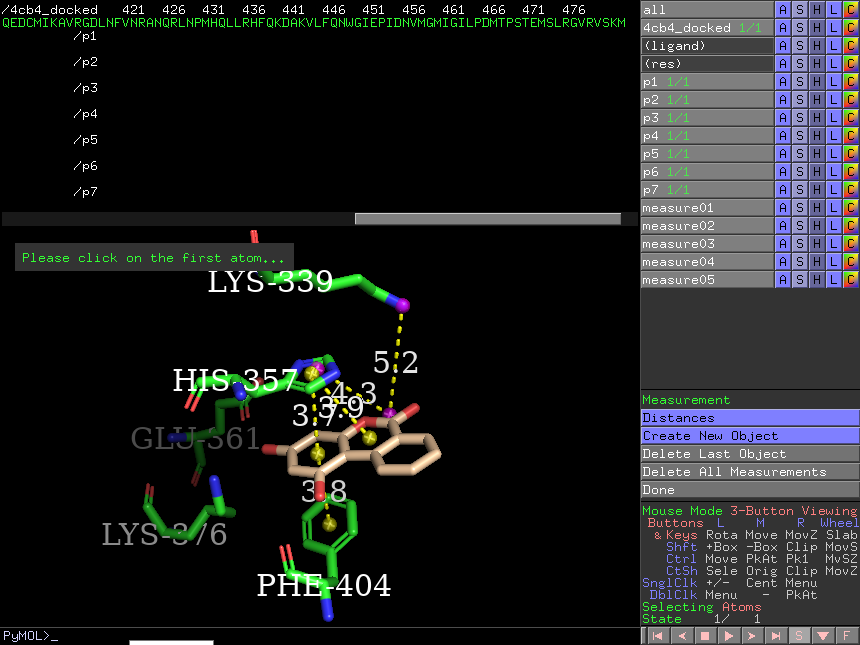
Wizard -> Measurement,点击创建的小球,两两一组,画出虚线

画出氢键和疏水作用

结果美化
命令行输入
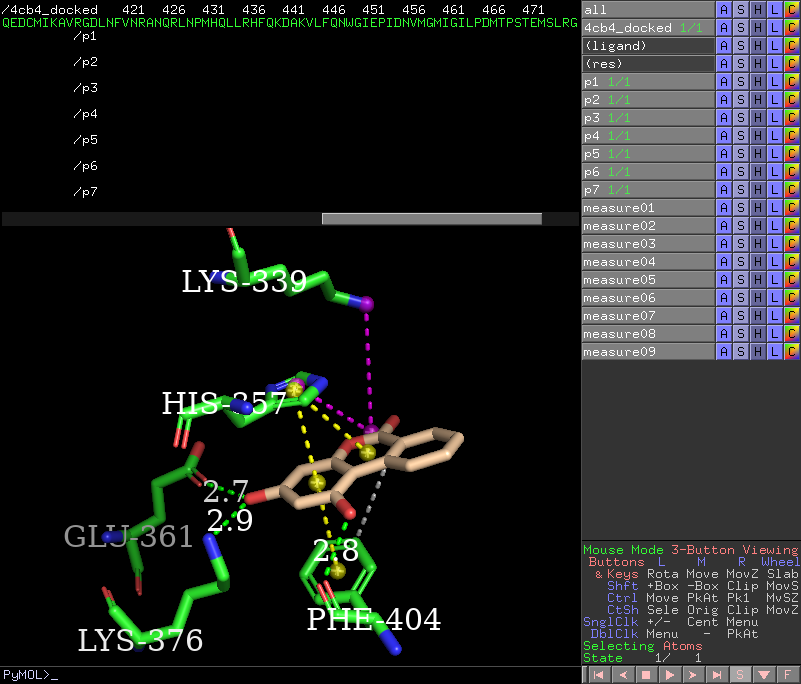
color magenta, measure04,color magenta, measure05将盐桥设为洋红色命令行输入
color green, measure06,color green, measure07,color green, measure08将氢键设为绿色命令行输入
color gray70, measure09将疏水作用设为70%灰色命令行输入
hide labels, measure*隐藏所有虚线上的标签命令行输入
show labels, measure06,show labels, measure07,show labels, measure08显示氢键上的标签

命令行输入
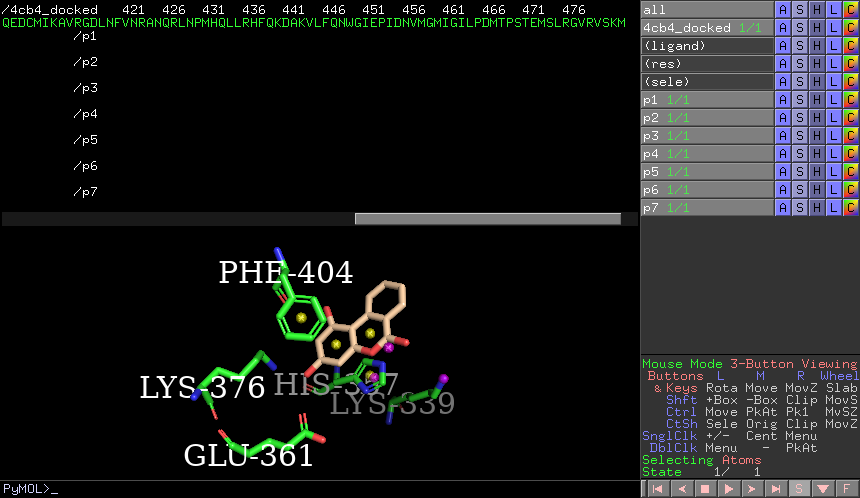
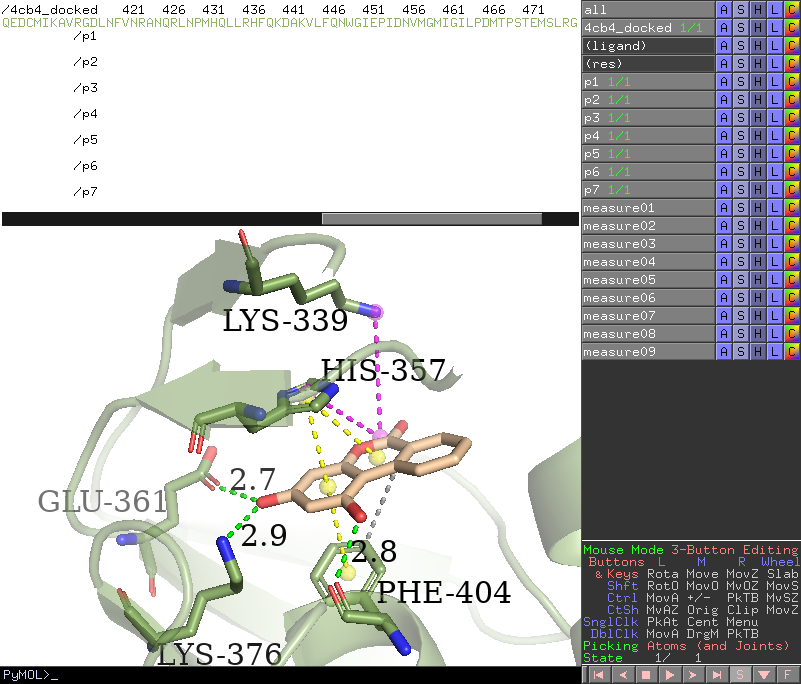
bg_color color将背景设为白色命令行输入
show cartoon显示蛋白卡通结构命令行输入
color smudge, !ligand & name C*设置除配体外的碳原子的颜色命令行输入
set cartoon_transparency, 0.5将蛋白卡通结构设为半透明,避免遮挡

图片保存
命令行输入
save focus.pse保存工作,防止意外,方便修改命令行输入
ray光线追踪,美化图片命令行输入
png focus.png, 16.93cm, 16.93cm, dpi=300保存图片,规格为16.93cm * 16.93cm,分辨率300dpi