文章信息
- 题目:Innovations to culturing the uncultured microbial majority
- 年份:2020
- 单位:瓦赫宁根大学
- DOI:10.1038/s41579-020-00458-8
- 杂志:Nature Reviews Microbiology (IF 34.209)
- 分类:Review
传统分离培养策略
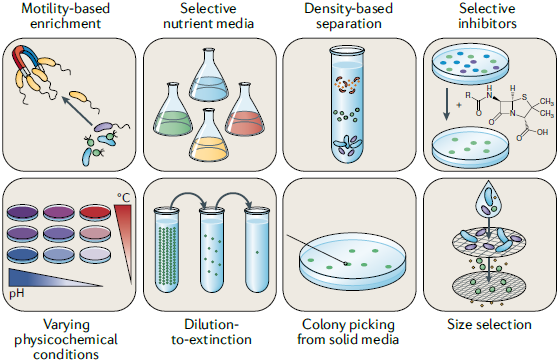
富集特定物种的技术:
设计选择性培养基(如:特定底物)
应用选择性理化条件(如:温度,pH,盐度,气相成分)
添加选择性抑制剂(如:抗生素,毒性化合物,代谢抑制剂)
添加或省略特定生长因子(如:氨基酸,维生素,金属离子)
筛选分离技术:
根据细胞大小进行过滤
基于质量的梯度离心
根据生长速度转接
极限稀释
根据趋向性筛选(如:趋光,趋氧,趋化,趋电,趋磁)
近期的成功培养案例
古菌
阿斯加德古菌超门(Candidatus Prometheoarchaeum syntrophicum)
目前已培养的与真核生物亲缘关系最近的古菌
使用一种新型生物反应器,富集12年
纳米盐古菌门(Candidatus Nanohaloarchaeum antarcticus)
- 结合经典富集方法与基于细胞大小的单细胞分选
Candidatus Argoarchaeum ethanivorans 和 Candidatus Ethanoperedens thermophilum
首次获得的与硫酸盐还原细菌互养的乙烷氧化古菌
传统选择富集方法
细菌
79个浮霉菌门的不同菌株
- 选择性富集,抗生素处理,固体培养基划线等传统方法
首个α-变形菌纲SAR11的淡水代表菌株(Candidatus Fonsibacter ubiquis)
- 寡营养培养基高通量极限稀释
TM7门(Saccharibacteria)的3个物种
与宿主放线菌一起分离自人的唾液样品
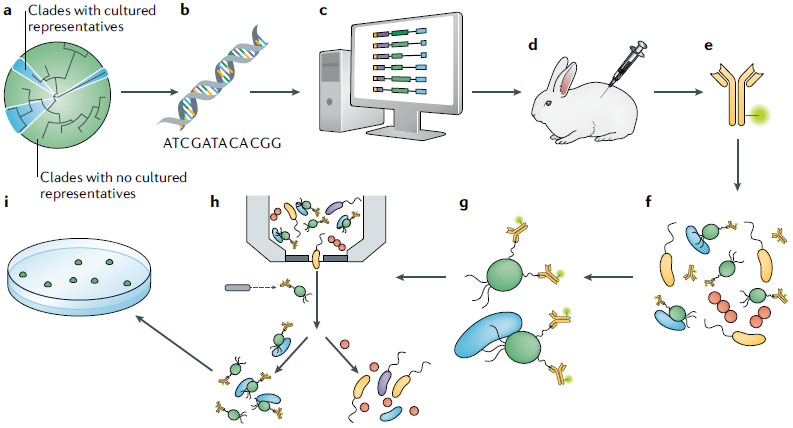
反向基因组学方法[1]
首个与β-变形菌互养的锰氧化细菌(Candidatus Manganitrophus noduliformans)
- 选择性底物富集加极限稀释
影响分离培养的因素
鉴定底物及生长条件
从休眠状态中复苏
共生依赖
物理接触或空间接近
理化环境条件
低丰度及竞争
即使目的微生物起初在样品中是富集的,在与快生长微生物共同富集培养过程中,其丰度也可能会因为竞争而迅速下降。近期的解决方法集中在从环境样品中分离单细胞进行接种,而不是从混合群落中逐渐富集[2,3]
新型培养技术
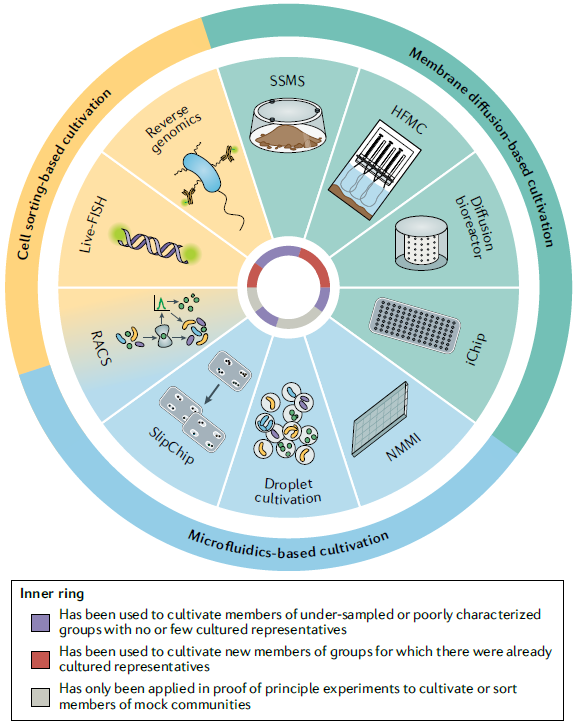
基于膜扩散的培养
由于难以复制天然生境的所有必须生长因子,使用孔径合适的滤膜将微生物细胞与环境样品形成物理分隔,但允许生长因子扩散到细胞周围[4]
中空纤维模腔装置(HFMC)[5]
i(isolation)Chip[2]
土壤底物膜系统(SSMS)[6,7]
大体积的扩散生物反应器[8]
微流控系统培养
通过细胞分选或物种/功能筛选进行分离
基于液滴或微流控的分选
显微光镊精确操纵和分离单细胞[12]
分离培养了纳米尺度的超嗜热古菌Nanoarchaeum equitans及其宿主Ignicoccus hospitalis[13]
流式细胞荧光分选(FACS)
荧光原位杂交(FISH)
近期有研究报道避免细胞固定及膜透化的”live-FISH”,并成功标记、分选并培养了天然海水中的α-变形菌,但细胞的存活率仍然非常低(1.24-2.82%)[14]
反向基因组学技术[1]

既能实现物种特异性标记和筛选,也能保持细胞的活性。根据宏基因组分箱获得的未培养微生物基因组,预测物种特异的带有胞外结构域的跨膜蛋白,以此作为抗原生产带有荧光标记的抗体。将抗体与环境样品进行孵育,标记目的细胞,通过流式细胞荧光分选收集,用于后续培养
基于拉曼光谱的单细胞分选(RACS)[15,16]
在有利于目标微生物发挥活性的条件下用重水进行培养,重水插入更活跃的细胞新合成的脂质,称为一种化学标签。拉曼显微镜配合微流控装置能够检测插入重水的细胞,用光镊精确分选
局限性
形成生物膜的微生物难以进行细胞分选
采集自环境样品的微生物带有非生物颗粒,干扰细胞标记及基于流动的方法
需要在分离前将细胞从样品颗粒上脱离下来[17]
新技术需要的大型仪器在厌氧环境中的操作适配性
难以为高通量的培养方法提供气态底物(如:氢气,二氧化碳,一氧化碳,甲烷)
高温培养条件下液体的快速蒸发
分离到单细胞只是第一步,还需要找到合适的培养基及理化条件维持目标微生物的生长
通过多组学数据预测目标微生物的表型特征,从而推测培养条件[18]。或者利用”next-generation”生理学方法,在单细胞水平上判断富集液或环境样品中目标微生物的代谢及生理特征[19]
筛选方法
直接观察
光学检测生长情况
PCR及基于测序的筛选
MALDI-TOF质谱
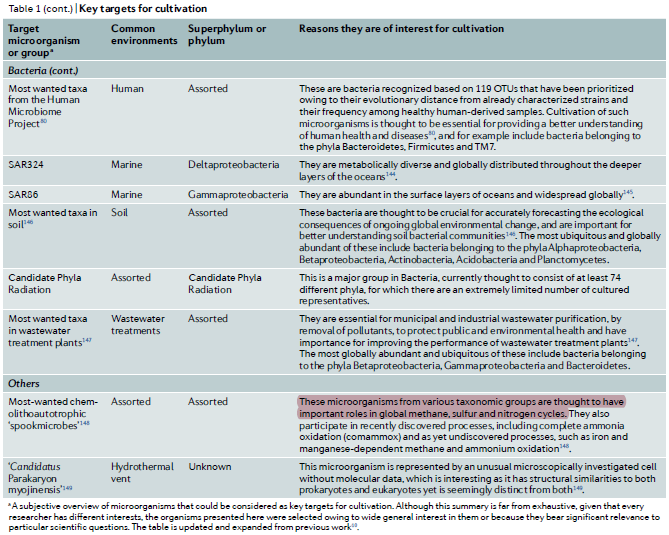
分离培养的目标
参考文献
[1] Cross KL, Campbell JH, Balachandran M, Campbell AG, Cooper SJ, Griffen A, Heaton M, Joshi S, Klingeman D, Leys E, Yang Z, Parks JM, Podar M. 2019. Targeted isolation and cultivation of uncultivated bacteria by reverse genomics. Nature Biotechnology 37:1314-1321.
[2] Nichols D, Cahoon N, Trakhtenberg EM, Pham L, Mehta A, Belanger A, Kanigan T, Lewis K, Epstein SS. 2010. Use of Ichip for High-Throughput In Situ Cultivation of “Uncultivable” Microbial Species. Applied and Environmental Microbiology 76:2445.
[3] Ge Z, Girguis PR, Buie CR. 2016. Nanoporous microscale microbial incubators. Lab on a Chip 16:480-488.
[4] Kaeberlein T, Lewis K, Epstein SS. 2002. Isolating “Uncultivable” Microorganisms in Pure Culture in a Simulated Natural Environment. Science 296:1127.
[5] Aoi Y, Kinoshita T, Hata T, Ohta H, Obokata H, Tsuneda S. 2009. Hollow-fiber membrane chamber as a device for in situ environmental cultivation. Appl Environ Microbiol 75:3826-33.
[6] Ferrari BC, Binnerup SJ, Gillings M. 2005. Microcolony Cultivation on a Soil Substrate Membrane System Selects for Previously Uncultured Soil Bacteria. Applied and Environmental Microbiology 71:8714.
[7] Svenning MM, Wartiainen I, Hestnes AG, Binnerup SJ. 2003. Isolation of methane oxidising bacteria from soil by use of a soil substrate membrane system. FEMS Microbiology Ecology 44:347-354.
[8] Chaudhary DK, Khulan A, Kim J. 2019. Development of a novel cultivation technique for uncultured soil bacteria. Sci Rep 9:6666.
[9] Ma L, Kim J, Hatzenpichler R, Karymov MA, Hubert N, Hanan IM, Chang EB, Ismagilov RF. 2014. Gene-targeted microfluidic cultivation validated by isolation of a gut bacterium listed in Human Microbiome Project’s Most Wanted taxa. Proceedings of the National Academy of Sciences 111:9768.
[10] Watterson WJ, Tanyeri M, Watson AR, Cham CM, Shan Y, Chang EB, Eren AM, Tay S. 2020. Droplet-based high-throughput cultivation for accurate screening of antibiotic resistant gut microbes. Elife 9.
[11] Eun Y-J, Utada AS, Copeland MF, Takeuchi S, Weibel DB. 2011. Encapsulating Bacteria in Agarose Microparticles Using Microfluidics for High-Throughput Cell Analysis and Isolation. ACS Chemical Biology 6:260-266.
[12] Wang X, Chen S, Kong M, Wang Z, Costa KD, Li RA, Sun D. 2011. Enhanced cell sorting and manipulation with combined optical tweezer and microfluidic chip technologies. Lab on a Chip 11:3656-3662.
[13] Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. 2002. A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont. Nature 417:63-67.
[14] Batani G, Bayer K, Böge J, Hentschel U, Thomas T. 2019. Fluorescence in situ hybridization (FISH) and cell sorting of living bacteria. Scientific Reports 9:18618.
[15] Berry D, Mader E, Lee TK, Woebken D, Wang Y, Zhu D, Palatinszky M, Schintlmeister A, Schmid MC, Hanson BT, Shterzer N, Mizrahi I, Rauch I, Decker T, Bocklitz T, Popp J, Gibson CM, Fowler PW, Huang WE, Wagner M. 2015. Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells. Proceedings of the National Academy of Sciences 112:E194.
[16] Lee KS, Palatinszky M, Pereira FC, Nguyen J, Fernandez VI, Mueller AJ, Menolascina F, Daims H, Berry D, Wagner M, Stocker R. 2019. An automated Raman-based platform for the sorting of live cells by functional properties. Nature Microbiology 4:1035-1048.
[17] Morono Y, Terada T, Kallmeyer J, Inagaki F. 2013. An improved cell separation technique for marine subsurface sediments: applications for high-throughput analysis using flow cytometry and cell sorting. Environmental microbiology 15:2841-2849.
[18] Gutleben J, Chaib De Mares M, van Elsas JD, Smidt H, Overmann J, Sipkema D. 2018. The multi-omics promise in context: from sequence to microbial isolate. Crit Rev Microbiol 44:212-229.
[19] Hatzenpichler R, Krukenberg V, Spietz RL, Jay ZJ. 2020. Next-generation physiology approaches to study microbiome function at single cell level. Nature Reviews Microbiology 18:241-256.